计算机学院王建新团队在基因组转座子检测和注释研究中取得新进展
来源:计算机学院 点击次数:次 发布时间:2024年07月09日 作者:胡康
本网讯 7月2日,国际权威期刊《自然通讯》(Nature Communications)在线发表了中南大学计算机学院王建新教授团队的最新研究成果“HiTE:一个基于动态边界调整方法的快速、准确全长转座元件检测和注释工具(HiTE:afast and accurate dynamic boundary adjustment approach for full-lengthtransposableelementsdetection andannotation)”。该论文提出了基于基因组组装数据的转座子检测和注释新方法,并开发了相应的软件HiTE。中南大学计算机学院胡康、倪鹏为论文共同第一作者,中南大学计算机学院王建新教授为论文共同通讯作者,中南大学为第一署名单位。本研究受国家重点研发计划、国家自然科学基金、湘江实验室揭榜挂帅项目等多个项目支持。
转座子(TEs)占据了大多数真核生物的重复区域,并已知对基因组进化和种内基因组多样性有显著影响。研究发现,TEs通过中断或调控关键基因在人体疾病和作物育种中发挥重要作用。近年来,基因组组装技术的进步极大改善了对转座元件进行全面注释的前景。然而,现有基于基因组组装的TE注释方法由于缺乏准确性和鲁棒性,仍需要大量的人工编辑。此外,即使是对已广泛研究的物种,目前可用的黄金标准TE数据库也不够全面,迫切需要一种自动化的TE检测方法来补充现有的数据库。
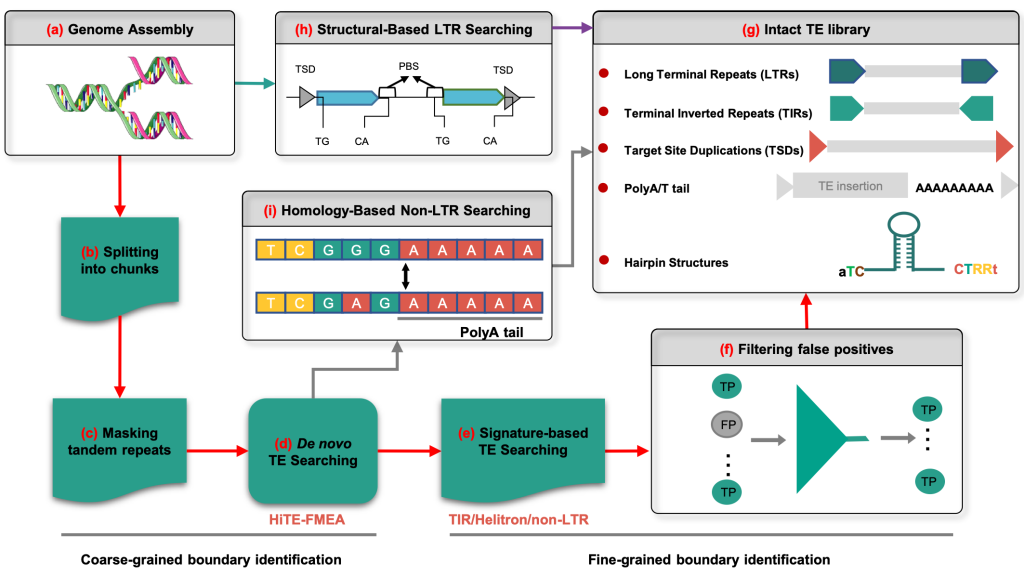
HiTE注释流程图
针对这一局限,王建新教授团队通过深入分析转座子元件的序列结构特征和生物复制原理,提出了动态边界调整算法,准确识别转座子元件的边界。在较为通用的95%评估阈值下,识别了远超已有工具的全长TE数量,例如在水稻中识别了1078个全长的TE序列,而已有最先进方法EDTA和RepeatModeler2分别只识别了506和378个。为了解决转座子识别中较高的假阳性难题,实现了多种高可靠的过滤算法,能够过滤绝大多数假阳性序列。实验结果表明,过滤后的HiTE获得了更多的全长TE数量,并且在几乎未降低敏感性的情况下,大幅度地提升了识别精度,并获得了93.56%的F1值,远超EDTA和RepeatModeler2的87.34%和54.82%。
(一审:戴寓欧 二审:邓皓迪 三审:李殷)









 分享:
分享: