计算机学院李敏教授团队发表与药学结合的跨学科突破性研究成果
来源:计算机学院 点击次数:次 发布时间:2025年06月07日 作者:朱慧敏
本网讯 中南大学计算机学院李敏教授团队前不久在国际顶级期刊《自然通讯》(Nature Communications)发表了一项突破性研究成果,题为“DeepDTAGen框架:一个统一的多任务学习框架,能够同时预测药物与靶标的结合亲和力,并生成针对特定靶标具有生物学意义的候选药物分子(DeepDTAGen:A Multitask Deep Learning Framework for Drug-Target Affinity Prediction and Target-Aware Drugs Generation.)”。计算机学院的来华留学博士研究生Pir Masoom Shah为论文第一作者。朱慧敏、卢长利、王凯丽为重要贡献作者。李敏教授为通讯作者,中南大学为第一署名单位。
药物发现是一个成本高昂且耗时漫长的过程。其核心目标之一是设计能够有效结合生物靶标(如酶、受体或信号蛋白)以产生治疗效果的分子。药物与靶标的结合强度称为结合亲和力,它直接影响药物的功效和安全性。然而,如何引导深度学习模型学习药物与靶标之间的结合关系,并利用该知识生成具有生物学意义的药物分子依然面临着巨大的挑战。传统的计算方法通常只侧重于预测药物-靶标亲和力或生成新分子,难以精确把握这两个任务之间内在的关联。
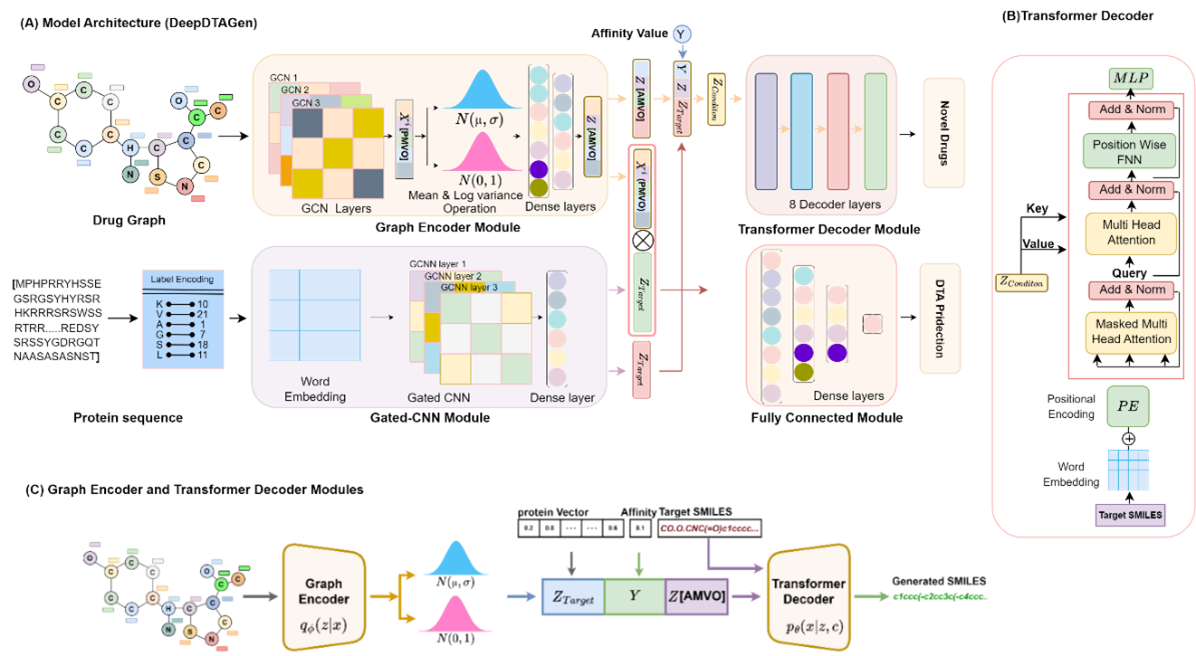
针对上述挑战,李敏教授团队开发了DeepDTAGen。这一新颖的多任务学习框架不仅能预测药物-靶标亲和力,还能在FetterGrad算法引导下,生成具有特定靶标活性的新颖药物分子。FetterGrad算法的核心作用在于实现跨任务的梯度对齐,确保模型在药物-靶标预测与分子生成任务中能够协调优化。
该模型提供两种模式生成SMILES:
1. SMILES模式(SMILES Method):该模式需要一个初始的SMILES字符串(代表现有药物分子)、靶蛋白特征以及期望的亲和力数值作为输入,其核心功能是基于目标亲和力优化现有的药物分子。
2. 随机生成模式(Stochastic Method):该模式仅需输入靶标信息和期望的亲和力数值,即可从头生成满足预期活性的新候选药物分子。
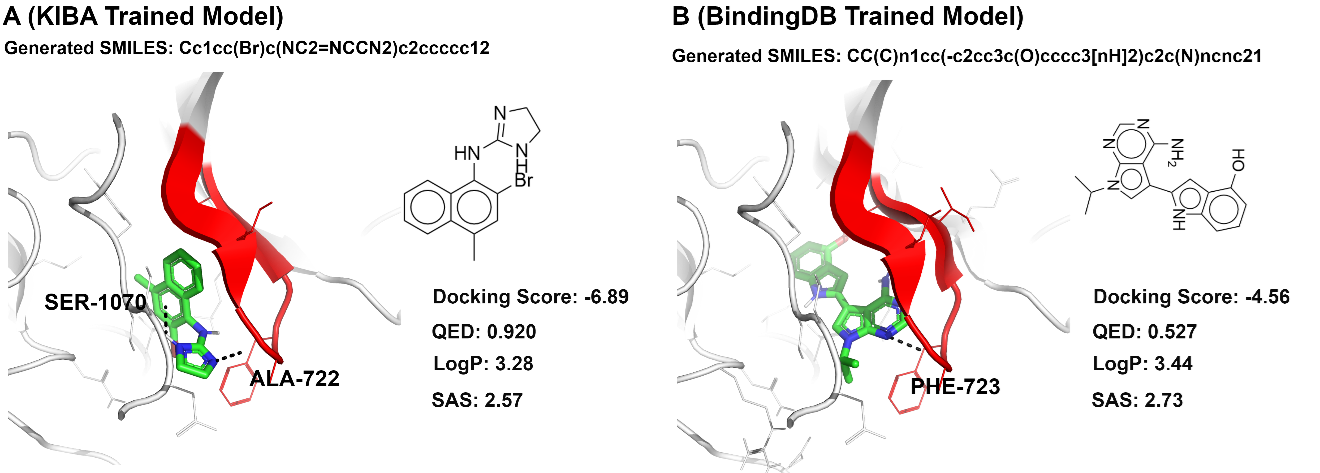
研究工作采用多种评估指标对DeepDTAGen进行验证,包括对接、利平斯基五规则(Lipinski’s Rule of Five),以确保生成的分子具备良好的类药性,同时针对EGFR(表皮生长因子受体)生成了候选药物分子,并通过分子对接实验验证生成的化合物能有效结合在该蛋白的活性位点上。

这项研究充分验证了DeepDTAGen作为多功能药物发现工具的潜力,展示了其生成新型候选药物分子的能力。通过整合亲和力预测与药物生成,DeepDTAGen为理解药物-靶标相互作用提供了新视角,为药物重定位和个性化医疗的应用铺平了道路。未来,团队计划将更多化学性质约束—如类药性(QED)和脂溶性(LogP)融入模型,并探索利用3D结构信息进一步提升框架性能。
(一审:张亚轩 二审:王轩 三审:李殷)









 分享:
分享: