中南大学李敏团队在蛋白质功能预测研究中取得重要进展
来源:计算机学院 点击次数:次 发布时间:2025年01月18日 作者:雷英琛
本网讯 近日,国际权威期刊《自然·通讯》(Nature Communications)在线发表了中南大学计算机学院李敏教授团队的最新研究成果“DPFunc:利用结构域引导的结构信息进行蛋白质功能精准预测(DPFunc: Accurately predicting protein function via deep learning with domain-guided structure information)”。该研究提出了一种基于深度学习的新方法DPFunc,有效整合蛋白质序列和结构信息,用于精准预测蛋白质功能。中南大学计算机学院王文康博士为第一作者,计算机学院的李敏教授为论文通讯作者,中南大学为第一署名单位。
蛋白质是生命活动的基本执行单元,精确解析蛋白质功能对于理解生物学机制和疾病治疗至关重要。随着高通量测序技术的快速发展,蛋白质序列数据的规模呈爆炸性增长,但已有的蛋白质功能注释仍远远滞后于数据积累速度。目前,仅有不到1%的蛋白质序列被系统性地注释了功能信息,这极大地限制了对蛋白质功能的深入理解和相关研究的推进。传统的实验方法虽然精准,但成本高、周期长,难以适应大规模蛋白质功能注释的需求。针对这一挑战,计算方法成为弥补实验注释不足的重要手段。然而,现有蛋白质功能预测方法普遍缺乏可解释性,难以揭示蛋白质结构与功能之间的关系,导致预测结果的准确性和解释性有限。尤其是在面对低同源性蛋白或新发现的蛋白质时,这些方法的预测效果大打折扣。
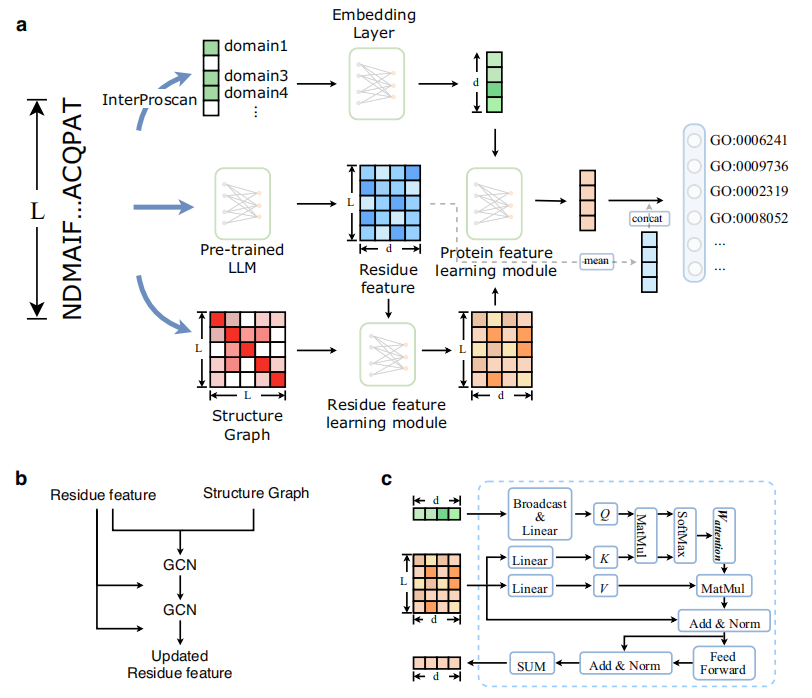
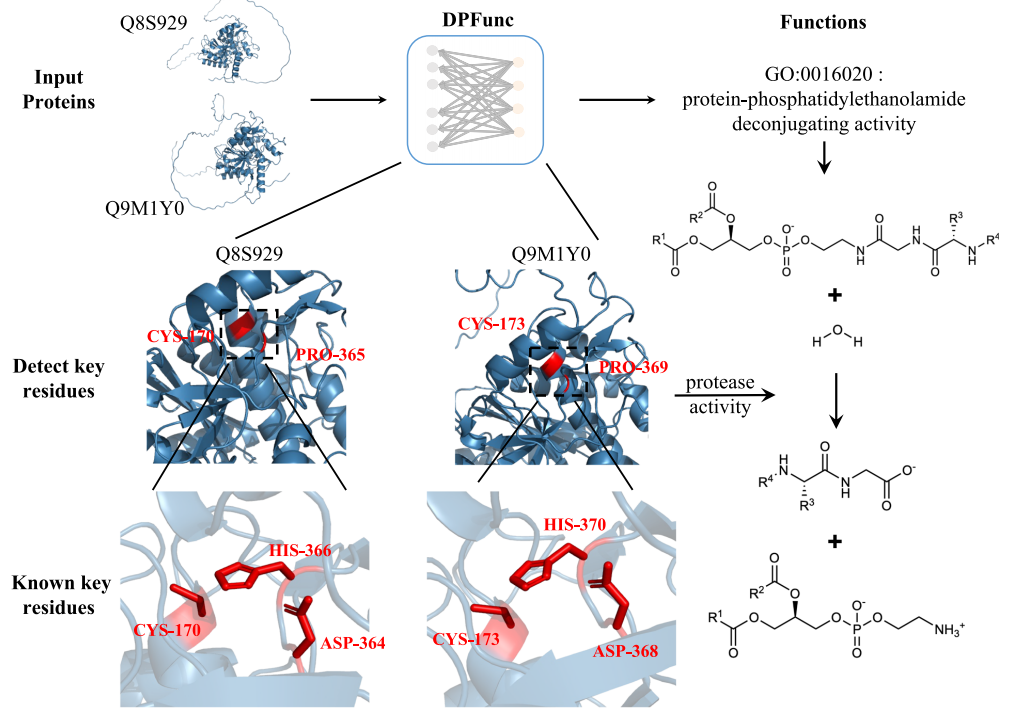
针对这一挑战,李敏团队创新性地提出DPFunc方法,通过融合蛋白质序列信息和结构域引导的结构特征,实现了蛋白质功能的精准预测。DPFunc能够识别蛋白质结构中与功能密切相关的重要区域,在多项基准测试中表现优异,显著超越现有的最先进方法。实验结果表明,DPFunc在蛋白质功能预测、关键残基识别和稀有功能检测等方面表现尤为突出。特别是在蛋白质结构功能关系分析中,DPFunc能够准确识别关键活性位点。例如,在半胱氨酸蛋白酶Q9M1Y0和Q8S929中,DPFunc成功检测到与功能密切相关的活性位点,表现出出色的解释性。此外,DPFunc在细菌功能注释中也展示出强大的潜力,在多种情况下显著优于其他预测方法,为大规模蛋白质功能预测和疾病机制研究提供了有力工具。
此次研究不仅推动了蛋白质功能预测领域的技术进步,也为理解蛋白质结构与功能的关系提供了新思路。未来,研究团队将进一步引入更多与蛋白质结构相关的生物学知识,推动DPFunc在动态功能预测和多功能关联研究中的应用拓展。
(一审:张亚轩 二审:王轩 三审:李殷)









 分享:
分享: